Im Labor wurden Versuche zur Replikation von RNA-Stücken gemacht. Manfred Sumper stellte im Jahr 1974 am Max-Planck-Institut für biophysikalische Chemie in Göttingen mehrere Versuche zusammen. Als Reproduktionsprotein fungierte die Qß-Replikase, das Replikationsenzym eines Virus, der Bakterien befällt. Zugegebene RNA-Moleküle vermehrten sich innerhalb deterministischer Grenzen, das heißt in genau bestimmten Zeitintervallen. Er machte allerdings die Entdeckung, dass in einem Reagenzglas, in das er keine Matrize gegeben hatte, ebenfalls RNA polymerisiert, das heißt neu gebildet wurde! Das erstaunliche an dieser De-novo-RNA war, dass sie einem Teilstück der Virus-RNA, die auch die Qß-Replikase codiert, sehr ähnlich war. So kamen in ihr die Tetramere CCCC und UUCG, ihre Komplemente und verstümmelte Trimere CCC und das GGG. Die Frage ist, ob das Enzym eine RNA synthetisiert hatte, nach der es selbst gebildet werden kann. In diesem Falle hätte man ein zentrales Dogma der Molekularbiologie verletzt, nach dem Informationen nur von den Nucleinsäuren (RNA) zum Protein (Enzym), nicht aber in umgekehrter Richtung fließen können.
Um diese Frage zu klären wurde ein weiteres Experiment gemacht: In ein Reagenzglas wurden die energiereichen Monomere der Basen Adenin, Uracil, Guanin und Cytosin und die Qß-Replikase gebracht. Eventuell vorhandene Matrizenmoleküle wurden zerlegt, sodass keine Messfehler auftreten konnten. Diese Lösung wurde auf mehrere Reagenzgläser aufgeteilt und dann wurde inkubiert. Inkubieren heißt die Temperatur für die Reaktion zur Verfügung zu stellen, quasi den Prozess zu starten. Die Inkubationszeiten waren für die einzelnen Reagenzien unterschiedlich lang, das kommt daher, dass die Neusynthese von RNA ein Zufallsbedingtes Phänomen und daher nicht vorherbestimmbar ist. Das Ergebnis war einleuchtend: In jedem Reagenzglas entstand nur ein einziges Produkt, aber die Produkte in den anderen Reagenzgläsern unterschieden sich sehr. Die Produkte hatten zwar einige Gemeinsamkeiten, doch diese sind vorerst ohne Belang. Die Reaktionsprodukte wurden nun ihrerseits auf mehrere Reagenzgläser aufgeteilt und der Prozess wurde wiederholt, man nennt das Serielle Übertragung. Es zeigte sich nach vielen solcher Übertragungen, dass die RNA evolvierte, das heißt sich an die Umgebungsbedingungen (Das Enzym Qß-Replikase) anpasste. Die bestangepasste RNA wurde also am schnellsten vermehrt, während die weniger gut angepassten RNAs langsam ausstarben, also nicht repliziert wurden. Am Ende gab es in allen Reagenzgläsern die nahezu gleiche RNA: Sie hatte eine Länge von 150 bis 250 Nucleotiden.
Bei mehreren Wiederholungen mit zum Teil anderen Bedingungen zeigte sich, dass es es RNA gab, die hohe Konzentrationen von Ribonucleasen, das sind Enzyme, die RNA an bestimmten Stellen in Stücke schneiden und sie damit evolutionär einen Schritt zurückwerfen, ertrugen. Sie waren so gefaltet, dass die Ribonucleasen nicht an die Schnittstellen herankamen und die RNA so nicht zerschneiden konnten. Wie wir wissen ergibt sich die Faltung der RNA aus ihrer Sequenz.
Das wichtigste an diesen Experimenten war, dass sogar die Moleküle der darwinschen Evolution unterliegen. Durch Fehler bei der Replikation (Mutationen) werden viele Varianten erzeugt, von denen nur die den Bedingungen bestangepasstesten überleben. Damit passt sich die Gesamtheit (Population) der RNA an ihren Lebensraum an. Erstaunlich ist auch, dass dieser primitive Vorgang, solch eine gute und schnelle Anpassung möglich macht.
Replikation ohne Enzym
Wenn man die Experimente mit solchen komplizierten Enzymen macht, stellt sich die Frage, ob dieser Evolutionsvorgang in der Ursuppe so ablaufen konnte. Durch zwei Aussagen kann man dies bejahen.
Erstens sind in der Ursuppe die Proteinoide enthalten, die eine schwache katalytische Wirkung haben. Lesen Sie dazu den vorangehenden Abschnitt.
Zweitens haben Experimente gezeigt, dass sich spontan kurze A-Oligomere, das sind Ketten von fünf bis zehn Nucleotiden, bilden, wenn man ein langes Polymer von Uracil-Nucleotiden in die Lösung bringt. Gibt man hierzu Blei-Ionen (Pb2+), steigt die Ausbeute an und die Monomere werden zu 75% korrekt über eine 5'-3'-Bindung verknüpft. Lesen sie hierzu bitte den Abschnitt Verknüpfungsprobleme. Gibt man hingegen zu Poly-C-Ketten A- und G-Nucleoside, dann erhält man Produkte, bei denen in 90% aller Fälle eine korrekte Basenpaarung erfolgt ist. Ich möchte hier wieder auf den Eigenbauplan der RNA verweisen. Nutzt man hier als Katalysator Zink (Zn2+), ergeben sich wesentlich längere Ketten mit einer Basenpaarungsgenauigkeit, die 20mal größer ist. Das erstaunliche hier ist, dass nahezu alle RNA-Polymerasen heute Zink-Ionen enthalten.
Leslie Orgel von Salk Institue for Biological Studies, der die oben genannten Experimente durchgeführt hat, fand auch heraus, dass Ketten, die viel Cytosin- und Guanin-Nucleotide enthalten, sehr viel stabiler sind als andere. Ihre Basen haften so gut aneinander, dass die Kopiergenauigkeit und auch die Translationsvorgänge wesentlich besser ablaufen konnten, als es die Ribosomen noch nicht gab.
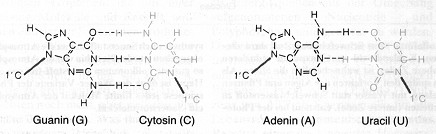
Durch kinetische und thermodynamische Untersuchungen erwies man, dass C-G-Bindungen 10mal stärker sind als A-U-Bindungen. Da Cytosin und Guanin sich über drei Wasserstoffbrückenbindungen verbinden und Adenin und Uracil nur über zwei, scheint das logisch. Die C-G-Bindung wirkt außerdem kooperativ auf benachbarte Nucleotide, dass heißt je mehr dieser Nucleotide nebeneinander liegen, desto stärker ist die Bindung der gesamten Kette. Aus diesen Erkenntnissen konnte man Modelle entwickeln und Strukturen berechnen, die zeigen, dass solche Strukturen, wie man sie heute bei der tRNA findet, Ergebnisse ebendieses Versuch-und-Irrtum-Prinzips sind. tRNA mittelt heute zwischen Aminosäuren und Basentripletts (Codons), indem sie an einer Seite das komplementäre Anticodon, auf der anderen Seite die entsprechende Aminosäure trägt. Sie besitzt eine typische Kleeblattstruktur.
Diese Experimente sollen uns zeigen, dass RNA auch ohne Enzyme zur Replikation fähig ist, aber die Proteinoiden sind an diesem Auslesevorgang sicherlich mit beteiligt.
Zurück | Vorwärts